费继峰教授PI团队
费继峰


费继峰
实验室负责人费继锋教授博士毕业于德国马克斯普朗克分子细胞生物学与遗传学研究所。长期从事大脑和脊髓的发育、损伤再生及基因编辑技术应用研究,建立了世界领先的以墨西哥钝口螈为模型研究中枢神经损伤修复的独特实验平台。近年来,主要关注于利用功能基因组学、单细胞测序、空间转录组学、活体成像和药物筛选等方法研究组织器官再生的分子和细胞机制,开发促进组织器官再生的小分子药物等。研究成果发表在Nature、Science、Nat Protoc、Nat Commun、PLoS Biol、PNAS、Cell Rep等学术期刊。主持国家重点研发计划国际合作项目、国家重点研发计划子课题、基金委国家自然科学基金重大研究计划培育项目、面上项目等多项研究项目。目前担任Medicine Advances副主编,Zoological Research编委等职务。
实验室实验面积充裕,实验室配备了开展常规及分子细胞生物学研究的仪器设备外,还配备超高分辨率激光共聚焦显微镜、光片显微镜等大型高端研究设备,以及全套大型蝾螈养殖系统,配套设备总价值超过2000万元,是国内目前规模最大的以蝾螈为主要模式生物研究发育与再生生物学的实验室。实验室与多个国际一流科研机构,如德国马克斯普朗克分子细胞生物学及遗传学研究所、德国德累斯顿再生治疗中心、奥地利分子病理学研究所、以及国内其它知名高校科研院所均有长期密切的合作研究,同时还与多家生物研究公司,如华大基因等,保持深度合作关系。
主要研究方向:
1.基于先进的显微成像技术,在体实时研究脊髓再生过程,重点探究关联细胞、关键细胞器、参与因子的动态作用机制;
2.利用多种脊髓损伤模型,研究启动脊髓再生的分子、细胞生物学机制;
3.利用脊髓损伤模型在体筛选促进脊髓再生的小分子药物;
4.利用单细胞测序、RNA测序、基因组学、蛋白组学等技术手段,筛选决定脊髓再生的关键因子;
5.研究脊髓再生过程中神经干细胞命运重编程及其机制。
实验室成员介绍:
费继锋 王鹍 乔娜 潘香羽 何子甜 李响 宋历 崔箫 易超 胡燕付苏雷王丽群 丘远辉
曾雁云 马惠 杨列可 闫曦文 李金曼 关婉琪 黄思琪 常兆强 林惠婷
实验室学术亮点:
中枢神经系统包括大脑的损伤后修复是目前医学及生物学领域的重大的前沿科学问题及难题。人类通常不能自发修复受损伤的中枢神经系统,并且目前缺乏有效的临床治疗手段。两栖类脊椎动物蝾螈(墨西哥钝口螈、Ambystoma mexicanum)具有强大的再生能力,其多种组织器官包括中枢神经系统、四肢等多种内外部组织器官在损伤后均可在细胞学水平及功能水平上完美再生修复。我们利用蝾螈为研究模型,以脊髓为研究对象,在细胞及分子生物学水平上探寻中枢神经系统损伤后的再生机制。为理解包括人类在内的哺乳动物脊髓损伤后修复的限制因素、重启哺乳动物脊髓损伤后细胞及功能水平上的修复提供可能的机制与可行的策略。
目前,我们已建立了基于CRISPR-CAS9基因编辑技术的蝾螈基因敲除、脊髓神经干细胞组织特异性基因敲除,以及定点基因敲入等技术。参与并完成了蝾螈全基因组、及多种组织器官在不同发育时期和再生阶段的高通量转录组的序列测定与组装。 构建了研究中枢神经系统损伤修复所需的转基因动物及疾病模型,并初步鉴定了中枢神经系统再生的相关基因。
代表性论文:
1. Pan XY, Zeng YY, Liu YM*, and Fei JF*. (2023) Resolving vertebrate brain evolution through salamander brain development and regeneration. Zoological Research, 18;44(1):219-222.
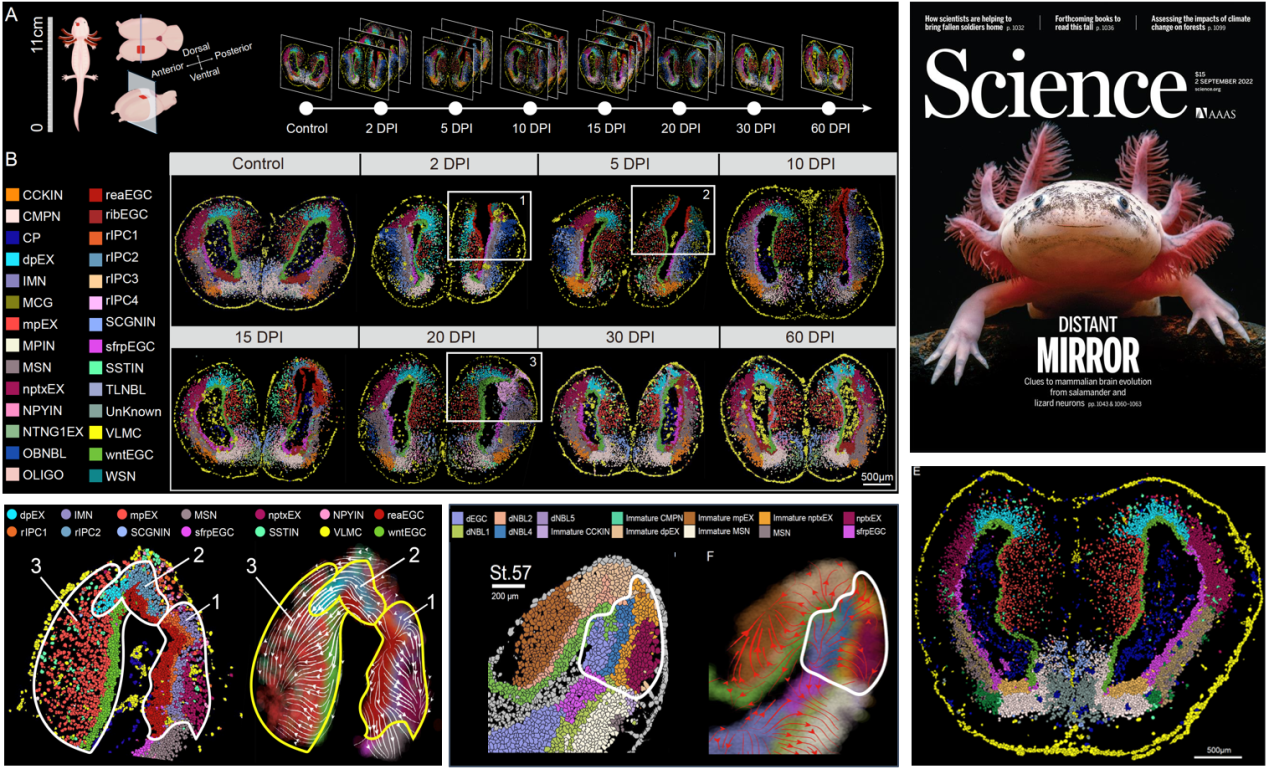
2. Wei X#, Fu S#, Li H#*, Liu Y#, Wang S#, Feng W#, Yang Y, Liu X, Zeng Y, Cheng M, Lai Y, Qiu X, Wu L, Zhang N, Jiang Y, Xu J, Su X, Peng C, Han L, Lou WP, Liu C, Yuan Y, Ma K, Yang T, Pan X, Gao S, Chen A, Esteban MA, Yang H, Wang J, Fan G, Liu L, Chen L*, Xu X*, Fei JF*, and Gu Y* (2022). Single-cell Stereo-seq reveals induced progenitor cells involved in axolotl brain regeneration. Science, 377(6610):eabp9444.(Cover story)
3. Ye F#, Zhang G#, E W#, Chen H#, Yu C#, Yang L#, Fu Y, Li J, Fu, S, Sun Z, Fei L, Guo Q, Wang J, Xiao Y, Wang X, Zhang P, Ma L, Ge D, Xu S, Caballero-Pérez J, Cruz-Ramírez A, Zhou Y, Chen M, Fei JF*, Han X* and Guo G*. (2022) Construction of the axolotl cell landscape using combinatorial hybridization sequencing at single-cell resolution. Nature Communications, 13(1):4228.
4.Fei JF*, Lou WPK, Knapp D, Murawala P, Gerber T, Taniguchi Y, Nowoshilow S, Khattak S, and Tanaka EM. (2018) Application and optimization of CRISPR/Cas9-mediated genome engineering in axolotl (Ambystoma mexicanum). Nature Protocols, 13(12):2908-2943.
5.Nowoshilow S#, Schloissnig S#, Fei JF#, Dahl A, Pang AWC, Pippel M, Winkler S, Hastie AR, Young G, Roscito JG, Falcon F, Knapp D, Powell S, Cruz A, Cao H, Habermann B, Hiller M*, Tanaka EM* and Myers EW*. (2018) The axolotl genome and the evolution of key tissue formation regulators. Nature, 554(7690):50-55.(Cover story) Highlighted in Nature, Nature Reviews Genetics and Current Biology.
6. Fei JF*, Schuez M, Knapp D, Taniguchi Y, Drechsel D, and Tanaka EM*. (2017) Efficient gene knockin in axolotl and its use to test the role of satellite cells in limb regeneration. Proc Natl Acad Sci U S A, 114(47):12501-12506.(Cover story)
7.Fei JF*, Knapp D, Schuez M, Murawala P, Zou Y, Drechsel D, and Tanaka EM*. (2016) Tissue- and time-directed electroporation of Cas9 protein-gRNA complexes in vivo yields efficient multigene knockout for studying gene function in regeneration. npj Regenerative Medicine, 1:16002. doi:10.1038/npjregenmed.2016.2
8. Wong FK#, Fei JF#, Mora-Bermúdez F, Taverna E, Haffner C, Fu J, Anastassiadis K, Stewart AF, and Huttner WB*. (2015) Sustained Pax6 expression generates primate-like basal radial glia in developing mouse neocortex. PLoS Biology, 13(8):e1002217. Highlighted in PLoS Biology.
9.Fei JF, Schuez M, Tazaki A, Taniguchi Y, Roensch K, and Tanaka EM*. (2014) CRISPR-Mediated Genomic Deletion of Sox2 in the Axolotl Shows a Requirement in Spinal Cord Neural Stem Cell Amplification during Tail Regeneration. Stem Cell Reports, 3(3):444-59. (Cover story)
10.Fei JF, Haffner C and Huttner WB*. (2014) 3' UTR-dependent, miR-92-mediated restriction of Tis21 expression maintains asymmetric neural stem cell division to ensure proper neocortex size. Cell Reports, 7(2):398-411.
实验技术亮点:
1)CRISPR/Cas9基因编辑技术
基因敲除方法
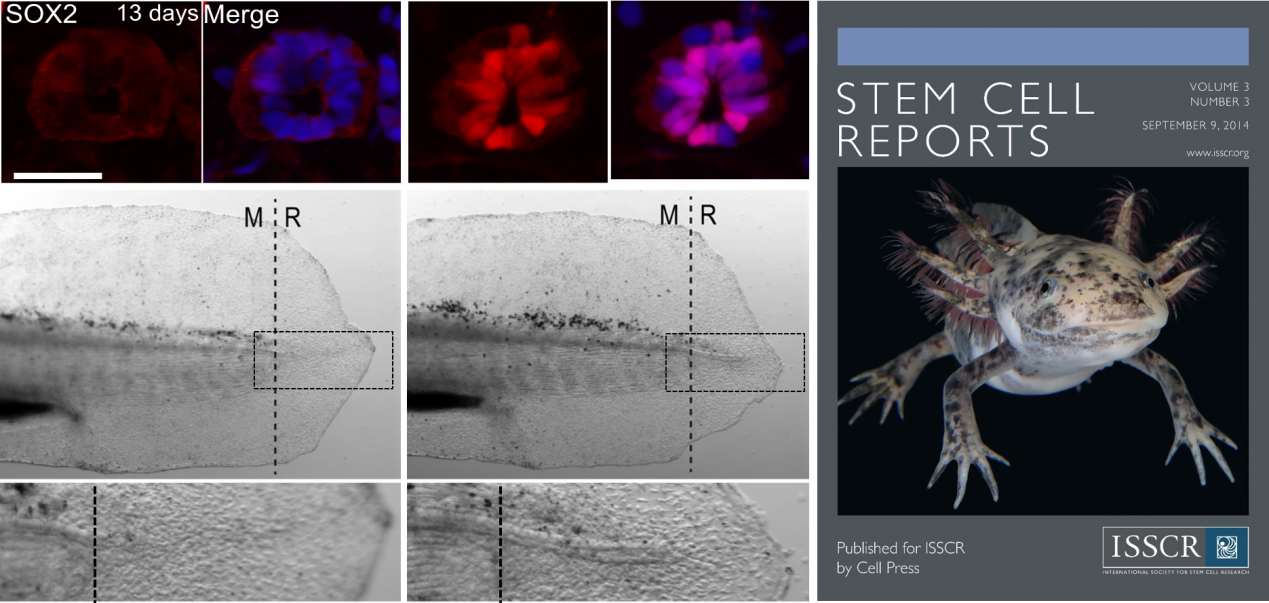
基因敲入方法
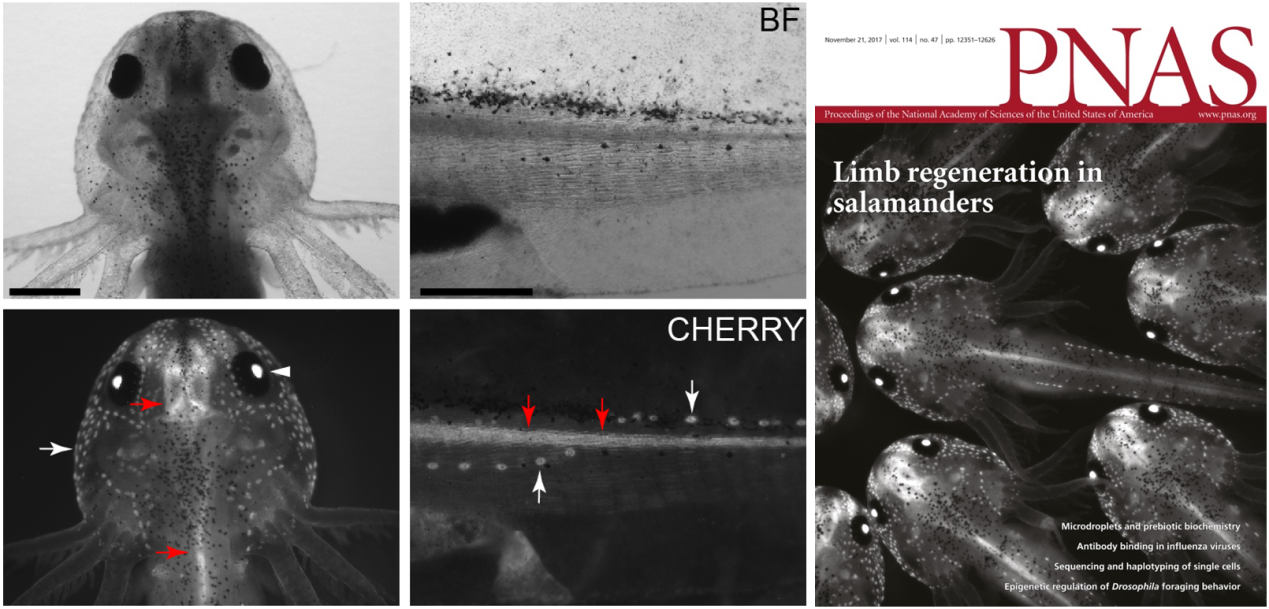
2)构建再生研究转基因动物模型

3)利用空间转录组技术研究组织器官再生机制
实验团队照片:




